乙型肝炎病毒RNA剪接的生物学意义和临床意义研究最新进展
介绍
乙型肝炎病毒(HBV)是一种嗜肝病毒,它有一个3.2kb的环形部分双链DNA,在受感染的肝细胞核内转化为共价闭合环状DNA(cccDNA)。HBV基因组包含四个重叠的开放阅读框(ORFs),编码乙肝e抗原(HBeAg)、核心蛋白、逆转录酶-聚合酶(P)蛋白、X蛋白和大、中、小表面蛋白。并通过单向转录生成前基因组/前核mRNA和三个亚基因组RNA。3.5kb双反子前基因组RNA(pgRNA)在基因组复制过程中充当封装模板执行反转录作用,并作为mRNA翻译出核心蛋白和P蛋白。前核mRNA稍长,其作用是编码HBeAg。2.4kbpreS1和2.1kbpreS2/S亚基因组mRNA编码三种表面蛋白,较小的0.7kbmRNA编码X蛋白[1]。
HBVcccDNA由细胞RNA聚合酶II转录而成,该酶已知可协同转录介导mrna的剪接、带帽和聚腺苷酸化。因此,我们认为5'端的“帽子”修饰了所有的病毒RNA。虽然HBV的RNA是在几个不同的启动子(前c/前基因组或基底核,S1,S2和X启动子)的控制下进行转录的,但它们都终止于一个共同的聚腺苷酸化信号(polyA),这一特殊现象生成了具有长链未翻译3'末端序列的成熟mRNA。此外,从HBV感染的人类肝脏组织和血清、HBV转基因小鼠和HBV转染的肝癌细胞系中[2],分别鉴定出了单一和多重剪接的HBV转录体pgRNA和pres2/smRNA。据报道,在体外实验中,高达80%的胞内核心颗粒和20%的胞外病毒颗粒包含经过剪接的基因组[3]。
乙肝病毒的持久存在,也被叫做隐性HBV感染/携带者(OBI),其特征是在受感染的肝细胞的细胞核中持续存在cccDNA,血浆和/或肝脏中HBVDNA水平非常低,使用最敏感的商品化试剂也检测不到乙肝表面抗原(HBsAg),核心抗体(抗-HBc)或表面抗体(抗-HBs)可有可无。OBI与HBV复制和病毒蛋白合成的强烈抑制有一定关联,涉及到病毒基因组的表达和/或细胞调控机制的复杂性,其机理目前尚不清楚。OBI可能诱发轻度持续的肝脏坏死炎症,这可能促使发展为肝硬化和肝细胞癌(HCC)[4]。因此,病毒RNA代谢的一系列特征,包括功能性前基因组和亚基因组RNA的加工、成熟、核输出等,将为病毒复制过程中重要分子机制的研究提供见解,并为未来开发新的抗病毒策略提供有价值的信息。
文中最新数据是针对之前HBVRNA剪接相关知识综述所做的补充[5]。
HBVRNA剪接过程
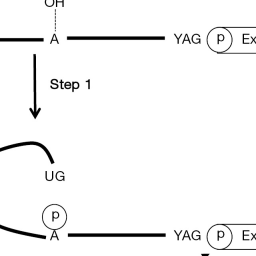
剪接是产生大多数功能性真核mRNA所必需的过程,这些mRNA通过剪接作用精确地去除内含子及连接外显子。这一成熟过程发生在细胞核中,在转录之后或与转录同时进行,并由多个异构核核糖核蛋白(hnRNPs)/RNA复合物介导,这些复合物被称为剪接体,在剪接位点以分步骤的方式进行组装。位于内含子内的剪接所需的基本序列元件是一个供体5'剪接位点(5'ss)、一个分支位点(包含一个关键腺嘌呤核苷酸)和一个受体3'剪接位点(3'ss),位于上游紧邻的多嘧啶片段可影响3'位点的功能(图1)。通过对真核细胞基因的分析,已经确定了一致性剪接信号序列,并研发了计算机程序来确定该序列[6,7]中假定的剪接位点位置。然而,预测的剪接位点经常被错误识别,这仍然是一个重要难题。尽管存在这些局限性,但在HBV基因组中仍然能够预测到类似的结构性和隐性的剪接信号,其数量和位置因基因型和单个毒株序列而异。
基于实验证据,目前已鉴定出16个pgRNA剪接变体[3,8-15]和4个preS2/SmRNA剪接变体[16,17](图2)。单、双或罕见的三重剪接变异体主要在体外转染细胞实验中发现,但也直接存在于不同HBV基因型感染者的肝组织和血清中。值得注意的是,除了在2350位置的3'ss位点外,在图2中报道的所有相应的供体和受体位点在预测程序的A-D基因型一致性序列中都被正确识别(未显示)。在患者样本和转染细胞中经常同时检测到来自pgRNA或preS2/SRNA的不同类型的剪接变体,这表明乙肝病毒前基因组和亚基因组RNA存在复杂的替代剪接模式。报道和研究最多的pgRNA剪接变体是两个2.2kb的剪接异构体:一个是在2447和489核苷酸之间的单剪接产物1(SP1),另一个则是在2447和2901核苷酸之间和2985和489核苷酸之间的双剪接产物[13]。SP1剪接变体被发现占转染肝癌源性细胞培养中pgRNA的25-30%,占患者样本中检测到的所有剪接变体的50-60%[3,11,13,15]。
pgRNA剪接的效率和剪接变异的性质因患者而异,从检测不到的剪接到广泛的、基因型/病毒株依赖的剪接[3,9,11,13,15]。产生这种变异的一个可能原因是,由于基于RNA扩增的检测方法的敏感性不足,检测不到那些罕见的剪接变体。未剪接的RNA和剪接变体通常在一个RT-PCR反应体系中共同扩增,存在竞争性,其显著差异可能影响最终的扩增效率。不同研究中使用的引物组合的特异性和扩增位点也可能影响检测结果。全病毒mRNA的扩增应优先于亚基因组的扩增。不同基因型和不同患者的序列变异也可能会造成、破坏或调节剪接信号序列的功能,从而影响到HBV剪接的定性和定量[18]。此外,一些研究从患者血清或细胞培养基中的病毒颗粒中分离出了缺陷基因组,从而确定了HBVRNA剪接的存在。在这种情况下,只能检测到保留RNA封装和逆转录所需的调控序列的剪接变体。因此,有可能低估了剪接的整体程度,因为一些剪接变异只存在于感染或转染细胞的细胞质中(如preS2/S剪接变体)。人肝癌细胞系Huh7和HepG2含有完全或部分复制HBV基因组的质粒,该类细胞的广泛应用也可能会导致一些偏差。与内源性HBV启动子相比,使用巨细胞病毒即早(CMV-IE)启动子来驱动病毒pgRNA或亚基因组RNA的转录时,观察到剪接的增加;与Huh7细胞相比,无论使用何种启动子,HepG2细胞中产生的剪接RNA水平都会更高(14;个人言论)。
HBVRNA剪接的调控
在转染的细胞培养、患者样本和不同的基因型或病毒株序列中观察到不同水平和不同形式的剪接体,表明HBVRNA的剪接不是一个结构性过程。将实际存在的剪接位点与预测的剪接位点进行比较,可以看出,一些已经存在的剪接位点不见得会被预测到(如图2中3'端2350位点),而一些预测的位点显然是不存在的。有趣的是,主要引起前S2/S序列剪接变体生成的5’端458位点、3’端1305位点和1385位点也存在于pgRNA序列中。然而,从未被报导过pgRNA的458-1305/85剪接变体或相应的封装缺陷DNA,尽管有研究假定剪接RNA可以保留功能性封装信号。此外,在大多数真核细胞中,pre-mRNA剪接与mRNA向细胞核外输出密切相关,它通过与剪接体蛋白直接作用,募集特定的输出因子,未剪接的RNA一般在细胞核内迅速水解。这个论点与部分剪接和未剪接HBVRNA的核质运输论点是相悖的。对于病毒的生命周期来说,大多数病毒RNA能够逃脱细胞固有的剪接机制并通过自身独立剪接机制进行输出,这是至关重要的。这表明,HBV剪接是由顺式作用元件和细胞和/或病毒反式作用因子精确调控的,这些因子控制着隐性剪接位点的激活和结构性剪接位点的沉默。
精确的选择性剪接不仅需要对5′端、3′端和分支点序列的识别,也需要外显子和内含子顺式调控元件[剪接调控元件(SREs)]募集序列特异性的RNA结合蛋白因子来激活或抑制邻近的剪接位点[19]。根据其位置和功能,通常把这些元件分类为外显子剪接增强子(ESEs)、外显子剪接沉默子(ESSs)、内含子剪接增强子(ISEs)和内含子剪接沉默子(ISSs)。通常,剪接增强子倾向于结合富含丝氨酸/精氨酸(SR)的蛋白,而剪接沉默子则用来募集hnRNPs。
在HBV基因组的1217-1582核苷酸处发现了一个被称为转录后调控元件(PRE)的顺式作用的调控元件。这种PRE存在于所有HBVmRNA的3'端。多项研究表明,PRE作为一个未剪接preS/SmRNA的重要的取向依赖性核输出元件,通过116个核苷酸功能性子元件(SEP1)募集TREX复合体,也可能维持了pgRNA稳定性和3'端的加工,但与核输出无关[2,20,21]。HBVPRE在HBV基因型中具有高度保守性,包含两个保守的二级结构元件(stem-loops),即HBVSLα和HBVSLβ,其功能尚未明确[22]。然而,位于1305位置的3’ss嵌在SLα中,有人认为该元件的二级结构可能会抑制该剪接位点从而允许未剪接HBVRNA向核外输出[23]。据报道,Pre-mRNA二级结构通过阻断或暴露剪接位点或顺式作用调控元件以及这些元件之间距离的空间修饰来影响剪接过程[24]。例如,相关的鸭肝炎病毒(DHBV)pgRNA剪接的调控依赖于病毒RNA的二级结构,该结构包含5'ss和3'ss,并拉近二者之间的距离[25]。
进一步的功能分析发现,在转染的培养细胞中,PRE不同元件能够正向和负向调节pgRNA剪接。剪接增强元件称为剪接调控元件1(SRE1),位于PRESLα和SLβ结构域上游(核苷酸1252-1288)[26],剪接沉默子位于PRE的3'区域(核苷酸1481-1585)[27]。这个沉默子原件折叠成一个双发夹结构,其正义链和反义链均强烈地抑制了与序列无关的选择性剪接。在PRE的上游也存在其他可能的顺式作用的调控元件。ZuPutlitz等人发现,在458位置5'ss和489位置3'ss之间的30个核苷酸缺失显著降低了pre/SRNA的表达[28]。此外,有报道称,在pgRNA的一个内含子剪接调控域内存在剪接激活元件(2951-2970和3051-3070核苷酸)和剪接抑制元件(3138-3143核苷酸)[29]。然而,这些数据存在过多体外实验中的人为干预,这些元件在HBV剪接的自然调节中的功能仍然不清楚。
假设剪接调控元件的活性是由反式作用因子调控的,而反式作用因子在很大程度上仍是未知的。假设细胞剪接调控蛋白(SF2/ASF,SC35SRp45和SRp55)会与SRE1结合,但缺乏确切的实验数据支持[2]。然而,已发现PTB相关剪接因子(PSF)可以强烈刺激pgRNA的剪接,很可能是通过直接与病毒RNA相互作用,特别是在PRE缺失的情况下[26]。在剪接的第二催化过程中,PSF与靠近3'ss的多嘧啶束相互作用,并可能取代3'剪接位点结合因子U2AF65[30]。此外,多项研究表明,另一种剪接调节蛋白——多嘧啶束结合蛋白(PTB)与PRE的3'端相互作用,能够抑制pgRNA剪接,并促进未剪接的PreS/SRNA向核外输出[26,31,32]。也有报道称,甘油醛-3-磷酸脱氢酶(GAPDH)能够绑定PRE,但这种绑定的功能意义尚不清楚[33]。
有一些证据表明HBV核心蛋白可能参与RNA剪接。核心蛋白穿过被感染细胞的核膜,并在包装过程中与pgRNA相互作用[34,35]。核心蛋白的羧基端序列构成一个非特异性的DNA和RNA结构域,该区域富含精氨酸残基,并包含三个丝氨酸磷酸化位点。这个羧基端序列的突变降低了优先被包装的剪接RNA的水平[36,37]。此外,有报道称核心蛋白的优先核定位与病毒RNA剪接的发生有关[38]。有趣的是,也有报道称DHBV核心蛋白的一小部分在接近剪接因子区间的核子域中也有积累[39]。综上所述,这些发现表明肝病毒核心蛋白可能在后转录病毒RNA代谢中具有非结构性的功能。
HBV剪接RNA编码的新蛋白质
到目前为止,从剪接的病毒RNA翻译出来的蛋白质中,有三种蛋白质已得到鉴定。在细胞裂解液、病毒粒子和亚病毒粒子中已经检测到由2447-2901位点剪接产生的43kDa聚合酶-表面融合糖蛋白(P-SFP)[40]。体外病毒复制不需要P-SFP,但在病毒成熟过程中它可以替代HBV长表面蛋白,推测该蛋白可能是参与病毒入侵的结构蛋白。另一项研究报道称P-SFP定位于内质网,并通过某种机制抑制HBV在转染细胞中的复制,其机制尚待研究[41]。有趣的是,从DHBVpgRNA中获得了剪接RNA编码的类似L蛋白,该蛋白在感染鸭肝细胞中的病毒复制中起重要作用,但在转染的DNA基因组中的病毒形成中没有起重要作用,并可能在病毒生命周期的早期阶段起重要作用[42]。
乙肝双剪接蛋白(HBDSP)是由2.2kb的双剪接pgRNA产生的,该pgRNA常见于慢性乙型肝炎患者和各种转染肝细胞衍生细胞系中[43]。有人认为HBDSP可能是一种多效激活蛋白,由激活蛋白-1-和CCAAT/增强因子结合蛋白结合位点介导,具有适度的反式激活活性。然而,HBDSP反式激活活性的确切机制、其生物学意义以及在HBV发病机制中的潜在作用尚不清楚。
SP1变体编码第三种已知的融合蛋白,称为HBV剪接生成蛋白(HBSP)[44]。这种包含93个氨基酸的蛋白质是由聚合酶的前46个氨基酸和剪接作用得到的新读码框中47个氨基酸融合而成的。已发现该蛋白在慢性HBV感染患者的肝组织中有表达。在HBV感染患者中已经发现存在HBSP抗体和HBSP介导的特异性T细胞应答,但在肝脏疾病中没有观察到[44,45]。有人认为,与HBV结构蛋白相比,HBSP的相对免疫优势可能是为病毒在慢性感染中持续存在提供了一种免疫诱饵机制[46]。体外实验中,HBSP的异位表达对病毒DNA的复制和转录没有影响,但可导致细胞凋亡[44,47]。HBV慢性携带者中HBSP抗体的存在也与病毒复制、肿瘤坏死因子α(TNFα)的升高以及肝纤维化的严重程度有关[48]。然而,另一项研究表明HBSP可以结合纤维蛋白原的γ链,并且抑制纤维蛋白聚合[49]。目前尚未发现HBSP在体内有导致细胞病变的作用,且其在HBV相关肝细胞癌的进展、侵袭和转移过程中所起的的作用尚未得到阐明。有趣的是,在体外HBSP过度表达和与组织蛋白酶B之间的相互作用增强了肿瘤诱导的血管生成,似乎激活了丝裂原激活蛋白激酶和Akt信号通路,并可能促进肝癌细胞的迁移和侵袭,从而促进了肝癌细胞的侵袭力[50]。最近,有报道称HBSP可以阻断TNF-α介导的信号通路从而参与先天免疫,并限制小鼠肝脏炎症的进展程度[51]。同样,据报道,慢性HBV感染患者血清中HBV剪接变体水平的升高与干扰素-α治疗产生的受损反应有关。在细胞和小鼠模型中,HBSP和去除N端的病毒聚合酶蛋白的表达能够导致IFN-α信号转导的强烈抑制[52]。还需要进一步的研究来确定这些可变剪接调节的病毒蛋白是否是该病毒为逃避先天和/或后天免疫而开发的额外策略的一部分。
在肝癌细胞中检测到了一种由2.2kb单剪接RNA(nt2447-nt489)编码的21.5kDa病毒蛋白(p21.5)[53]。据报道,该蛋白(一种比前核心/核心蛋白短的单氨基酸)的表达能够干扰核衣壳的形成,可能导致慢性感染患者中单剪接RNA的升高,后者在HBV复制的反馈调节中发挥相应的功能。在这种情况下,在体内和体外,从单剪接RNA翻译的p21.5水平的增加可能会减少核衣壳的生成,从而有助于病毒持久性存在。
通过检测多个剪接RNA编码的蛋白质,提示了在HBV感染过程中病毒RNA选择性剪接具有潜在功能,需要更多的研究来确定其他假定的新病毒蛋白,并阐述少数已知的功能。然而,不仅关注重排外显子的编码能力,还要关注内含子作为合适的复合体而不仅仅是剪接废物的编码能力。最近高通量基因表达分析方面的改进显示,非编码RNA(ncRNA)在人类转录组中占绝大多数,它们在基因表达中的调节作用已被广泛认可。有趣的是,已经发现非降解的长内含子ncRNA被选择性地输出到细胞质中,并参与整体基因表达调控,要么直接充当反式作用的调控元件,要么充当较短的调控microRNA(miRNA)的前体[[54]审稿中]。病毒编码/非编码RNA与宿主mRNA和miRNA之间的相互作用已有文献报道[55]。在HBVRNA中,一些与宿主miRNA互补的位点(如miR-122,miR-15a/16簇)参与了病毒基因表达、病毒复制和肿瘤抑制的调控[[55]审稿中]。这些互补位点像海绵一样结合和隔离宿主miRNA,可能有助于病毒持续感染和肝细胞癌的发展。有趣的是,最近的一项研究表明,转染人肝癌细胞和HBV转基因小鼠中,2.2双剪接RNA的表达显著降低了HBVpgRNA和XRNA的水平[56]。2.2双剪接RNA通过与诱导应激颗粒形成的TATA结合蛋白(TBP)相互作用,能够抑制病毒转录。这些数据表明,剪接RNA可能作为一种抑制性非编码RNA,调节HBV复制。需要进行更多的研究来确定其他HBV剪接RNA和/或内含子ncRNA是否参与RNA相关的调控过程,是否影响慢性乙型肝炎的发展过程。
HBV感染过程中的剪接
乙型肝炎病毒感染患者剪接变异体的高患病率[无症状HBsAg+献血者中约50%[15,57]至>90%轻度活动性肝炎[12]或肝癌[58]]和HBV序列中剪接位点的序列保守性强有力地支持了剪接RNA和相应假定编码蛋白的功能。在鸭和土拨鼠肝炎病毒中也发现了RNA剪接体,这表明在肝病毒进化过程中存在一些强大的功能性机制来保留剪接的能力[25,42,59]。然而,剪接也能破坏HBV编码病毒结构蛋白的开放性读码框,剪接位点的突变表明,体外病毒复制中不需要pgRNA剪接体[9,11]。
经过剪接的pgRNA包装衣壳后生成了含有缺陷病毒DNA基因组的病毒颗粒[3,10,12,13,15]。剪接和未剪接基因组在同一肝细胞内共存有利于其互补和从受感染细胞中释放剪接变体。在共转染实验中,已证明全病毒基因组产生的病毒蛋白可以增强pgRNA双剪接变体的复制,且无需复制未剪接的基因组[58]。据报道,某些有缺陷的HBV基因组的功能类似有缺陷的干扰颗粒,通过削减野生型病毒(像描述的其他病毒)的复制,从而提高病毒持久性[56,60]。相反,另一项研究报道,在共转染HepG2细胞时,2.2kb的双剪接变体增强了和其匹配的3.2kb全HBV基因组的复制以及其他来自B和C基因型中分离出来的异源全基因组的复制[58]。
血清盘中剪接病毒DNA的测序结果表明,在HBV感染的早期阶段,只有部分个体的剪接体检出时间与HBsAg检出时间相一致或略有延迟[15]。在另一项研究中,preS2/SmRNA剪接体似乎直接影响了HBsAg的检出限[16]。这些数据表明,与高传染性病毒的血清窗口期相比,HBsAg的检测能力和伴随的低传染性可能与pgRNA和/或preS2/SmRNA剪接的启动有关。在确定HBV感染剂量时,应把循环剪接变体的存在和程度考虑在内[15]。
在慢性乙肝表面抗原阳性患者中也发现有剪接变体的存在,而在那些患有急性自限性临床病症的患者中却很少发现[3,10,12,13]。无论如何,应该慎重对待这些观察结果,因为HBV剪接体的检测能力可能跟所检测的病毒核酸的性质(受感染肝细胞中的病毒RNA或DNA相对于血清中循环的包装衣壳的DNA)有所不同,且受上述分子检测能力的影响。此外,剪接变异体的相对数量会因患者和不同的基因型而异[3,15,57],使用标准化检测方法对个体病例进行贯序研究可能更好地描述这些剪接变异体的临床意义。然而,据报道,SP1变体在肝脏活检中的表达与低组织学活性指数相关[38],HBVRNA剪接体的减少与复杂的HBV变异性和进展为肝硬化相关[61]。其他研究表明,2.2kb剪接DNA在转染的Huh7细胞中表达,导致核心蛋白在细胞核和细胞质中显著积累,以及HBeAg分泌增加,这可能有助于免疫耐受形成[12,38,44]。这些观察结果支持HBVRNA剪接可能优先发生在慢性HBV感染的早期阶段的观点,以及剪接变异的免疫调节功能可能有助于HBV持久性的假设。
相反,其他研究者报道了在肝细胞癌患者的肿瘤组织中存在剪接变体[62]。据报道,2.2kb剪接变体的相对含量在肿瘤组织中高于在肿瘤周围组织中[62]。在另一项研究中,在严重肝坏死和纤维化患者中,剪接HBVDNA相对于总HBVDNA(剪接+未剪接)的平均比率较高[63]。这些观察表明剪接变体可能在肝细胞癌发展的发病机制中发挥作用。已经证明2.2kb剪接变体能增强细胞培养中全HBV基因组的复制,而增强效果需要有X基因的表达[58]。公认X蛋白是一种能激活病毒基因和致癌基因的转激活蛋白[64]。理论上,除preS2/S外,所有已报道的剪接变体都保留了潜在的功能性增强子I/X启动子和X基因序列。有假说认为剪接变异体携带的X基因的复制效应的增强可能促使HBV在肝细胞癌患者人群中的持续复制中发挥作用[58]。
最近有报道称,乙肝病毒整合进入细胞周期蛋白A2基因的内含子后,哺乳动物的剪接机制利用HBV基因组中nt282和nt458的外源剪接位点产生一个假外显子,形成一个HBV-cyclinA2嵌合转录体,这种现象出现在12.5%的肝细胞癌患者中,但在经检测的非肝癌相关的HBV肝硬化样本[65]中却没有发现。融合嵌合蛋白具有不可降解的特性,并促进了细胞周期的进展,显示了其潜在的致癌功能。哺乳动物在pre-mRNA剪接机制引导下,HBV基因组整合形成的HBV-人嵌合转录体可能具有潜在的致瘤作用,并参与HBV相关肝细胞癌的发展。
HBV剪接和隐匿性感染
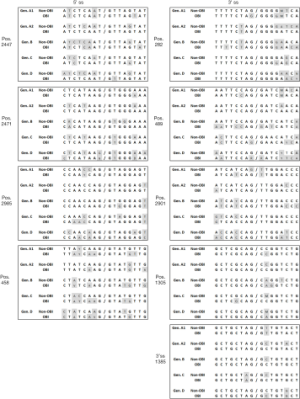
HBVpgRNA的剪接体已经在HBsAg阳性的慢性感染者中被广泛研究,且与总病毒载量相关[15]。很容易推测的是,这种机制可能与OBI的某些特定方面有关,比如明显有限的病毒复制。然而,目前还没有有关OBI中pgRNA剪接频率的数据。OBI携带者血液中的HBVDNA载量通常极低,标准的扩增方法可能缺乏一定的敏感性,需要适当的方法来检测衣壳内的未剪接和已剪接的基因组。根据我们的经验,在大多数OBI样本中,很少能获得完整的基因组,只有部分基因组序列(400-1200个核苷酸)被成功扩增[66]。在体外转染实验中,人们对评估HBV菌株的复制特性很少有兴趣。此外,由于伦理原因,没有临床表现的OBI携带者的肝脏活检调查很难获取。一般来说,普遍认为最小的pgRNA剪接位点在OBI序列中相比于非OBI序列显得保守,这表明在大多数情况下,OBI病毒RNA可能保留了进行剪接的能力,而与基因型无关(图3)。然而,某些基因型中一些特定剪接位点的序列变异可能会影响剪接:OBI基因型D序列5'ss的2471位点和3'ss的489位点,OBI基因型B序列3'ss的489位点,OBI和非OBI基因型A1序列的3'ss的2901位点(图3)。序列变异主要发生在经典的剪接位点附近,但与OBI无明显关联,除了5'ss458位点附近的OBI序列(图3)。RNA二级结构对于适当暴露剪接序列和招募剪接蛋白至关重要[67]。鉴于有许多例子表明RNA结构有助于剪接,因为有许多结构会抑制剪接,特定的突变可能会引起RNA结构的变化,从而导致不同的剪接结果。此外,通常观察到的OBI基因组的高度遗传变异性可能对一些剪接调控元件产生负面影响,这种可能性不能排除[17]。
为了研究OBI携带者经历病毒再激活过程中HBV突变的功能作用,本文描述了一个转录后机制影响到HBsAg表达的单一突变[16]。基因序列分析和位点定向诱变(SDM)结果表明,S基因中5′ss458位点单个G→A的替换(G458A)可诱导HBsAg表达下调,抑制preS2/SmRNA剪接。矛盾的是,剪接抑制能够导致preS2/SmRNA及其翻译产物的功能显著下降。作者推测,G458A突变可能通过破坏RNA推定的5'ss二级结构,改变preS2/SRNA和外显子结合蛋白之间的相互作用(图4),从而调节转录后RNA的加工过程。已经证实,RNA的结构可以隔离碱基对结构中的5'ss、3'ss或分支点,从而抑制剪接功能,这类似于一个HBV固有的preS2/S5'ss的假设结构[67]。然而,该结构是使用计算机建模程序MFOLD预测的,并且尚未通过生物化学方法或通过测试补偿突变来验证还原这个结构。另一个需要记住的问题是,剪接可能在慢折叠RNA结构形成之前发生共转录,以及RNA折叠和蛋白质结合到单链RNA构象之间可能存在的竞争[67]。

虽然在另一个OBI病毒株中尚未报道G458A突变,但是在pre-S2/S5'ss的458位点附近观察到的变异频率明显高于非OBI病毒株,而在这两种类型的病毒株中3'ss的1305和1385位点相对保守,与HBV基因型无关(图3)。在最近报道的献血者中,B型OBI中的25/55(45%)和C型OBI中的14/33(42%)表现出了5'ss458位点附近存在突变,而非OBI中B型和C型的突变位点分别为5/47(11%)和5/48(10%)。预测基因型B和C的非OBI序列像之前描述的那样以相同的茎环二级结构进行折叠(图4)[16,17]。在OBI变体中,44%的B型OBI和36%的C型OBI异质剪接供体突变破坏了这种茎环结构,与G458A突变类似。最近也有报道类似的干扰性突变发生在4.5%的OBI患者乙肝病毒序列中[68]。可以预见,非OBI变异体找不到类似的替代结构对茎环结构产生影响。通过少数OBI菌株和非OBI对照菌株中得到表达S编码区的质粒,瞬时转染huh7细胞,结果表明,一些干扰性突变确实能降低剪接效率,但与HBsAg分泌水平无关[17]。据推测,扩增剪接RNA产物含量与HBsAg分泌水平之间缺乏相关性,可能是因为剪接RNA短于非剪接RNA,更能有效得到扩增。然而,还需要更多的研究来调查影响preS2/S3'ss和/或相应的分支点位发生突变的影响,以进一步证明剪接作用和HBsAg分泌之间存在直接的功能性联系。
目前尚不清楚突变可能影响preS2/S5'ssd结构的突变或剪接本身是否直接对HBsAg的生成产生负面影响,需要开展更多针对OBI菌株的深度研究。然而,有一些证据强烈表明,5'ss的458位点是维持preS2/S高度转录水平和HBsAg生成所必需的调控元件一部分。剪接本身可能不直接参与,但众所周知,RNA的剪接与RNA的核输出密不可分。因此可以假设,在这种假定的调控元件中,包括G458A在内的突变可能会影响剪接体形成时RNA-蛋白质的相互作用,从而导致核输出减少或RNA稳定性降低。然而,HBsAg表达下调后的转录机制及使HBsAg难以检测等机制造成了隐性HBV携带者更具有特征性,其作用机理仍未阐明[17,69]。
展望
针对乙肝病毒RNA的剪接体的研究已经牢固树立,并且对于相对较小病毒基因组表达水平的复杂性研究方面也有所进展。剪接位点的高度保守、感染过程中剪接调控的复杂性以及剪接病毒RNA编码的独特蛋白质的存在,都强烈暗示了剪接机制在HBV生命周期中起到的功能性作用。我们还需要进一步的研究来确定宿主因子、其他顺式作用元件、RNA-RNA、RNA-蛋白质和蛋白质-蛋白质之间的相互作用,这些相互作用参与了HBVRNA剪接的复杂调控机制。了解HBV剪接的调控可能为其在隐匿携带者中长期存在HBV提供有价值的线索。
Acknowledgments
Funding: None.
Footnote
Conflicts of Interest: Both authors have completed the ICMJE uniform disclosure form (available at http://dx.doi.org/10.21037/aob.2017.05.01). DC serves as an unpaid editorial board member of Annals of Blood from Dec 2016 to Dec 2019. JPA serves as an unpaid editorial board member of Annals of Blood from Dec 2016 to Dec 2018. The authors have no other conflicts of interest to declare.
Ethical Statement: The authors are accountable for all aspects of the work in ensuring that questions related to the accuracy or integrity of any part of the work are appropriately investigated and resolved.
Open Access Statement: This is an Open Access article distributed in accordance with the Creative Commons Attribution-NonCommercial-NoDerivs 4.0 International License (CC BY-NC-ND 4.0), which permits the non-commercial replication and distribution of the article with the strict proviso that no changes or edits are made and the original work is properly cited (including links to both the formal publication through the relevant DOI and the license). See: https://creativecommons.org/licenses/by-nc-nd/4.0/.
References
- Seeger C, Mason WS. Hepatitis B virus biology. Microbiol Mol Biol Rev 2000;64:51-68. [Crossref] [PubMed]
- Sommer G, Heise T. Posttranscriptionnal control of HBV gene expression. Front Biosci 2008;13:5533-47. [Crossref] [PubMed]
- Sommer G, van Bömmel F, Will H. Genotype-specific synthesis and secretion of spliced hepatitis B virus genomes in hepatoma cells. Virology 2000;271:371-81. [Crossref] [PubMed]
- Pollicino T, Saitta C. Occult hepatitis B virus and hepatocellular carcinoma. World J Gastroenterol 2014;20:5951-61. [Crossref] [PubMed]
- Candotti D, Allain JP. Biological and clinical significance of hepatitis B virus RNA splicing: a role for spliced variants in occult infection?. In: Allain JP, Fu Y, Li C, et al. editors. Occult hepatitis B infection, 1st ed. Beijing: Science Press, 2015:90-107.
- Wang M, Marin A. Characterization and prediction of alternative splice sites. Gene 2006;366:219-27. [Crossref] [PubMed]
- Dogan RI, Getoor L, Wilbur WJ, et al. SplicePort-An interactive splice-site analysis tool. Nucleic Acids Res 2007;35:W285-91 [Crossref] [PubMed]
- Chen PJ, Chen CR, Stung JL, et al. Identification of a double spliced viral transcript joining the separated domains for putative protease and reverse transcriptase of hepatitis B virus. J Virol 1989;63:4165-71. [PubMed]
- Su TS, Lai CJ, Huang JL, et al. Hepatitis B virus transcript produced by RNA splicing. J Virol 1989;63:4011-8. [PubMed]
- Terré S, Petit AM, Bréchot C. Defective hepatitis B virus particles are generated by packaging and reverse transcription of spliced viral RNAs in vivo. J Virol 1991;65:5539-43. [PubMed]
- Wu HL, Chen PJ, Tu SJ, et al. Characterization and genetic analysis of alternatively spliced transcripts of hepatitis B virus in infected human liver tissues and transfected HepG2 cells. J Virol 1991;65:1680-6. [PubMed]
- Rosmorduc O, Petit AM, Pol S, et al. In vivo and in vitro expression of defective hepatitis B virus particles generated by spliced hepatitis B virus RNA. Hepatology 1995;22:10-9. [PubMed]
- Günther S, Sommer G, Iwanska A, et al. Heterogeneity and common features of defective hepatitis B virus genomes derived from spliced pregenomic RNA. Virology 1997;238:363-71. [Crossref] [PubMed]
- Abraham TM, Lewellyn EB, Haines KM, et al. Characterization of the contribution of spliced RNAs of hepatitis B virus to DNA synthesis in transfected cultures of Huh7 and HepG2 cells. Virology 2008;379:30-7. [Crossref] [PubMed]
- El Chaar M, El Jisr T, Allain JP. Hepatitis B virus DNA splicing in Lebanese blood donors and genotype A to E strains: implication for hepatitis B virus DNA quantification and infectivity. J Clin Microbiol 2012;50:3159-67. [Crossref] [PubMed]
- Hass M, Hannoun C, Kalinina T, et al. Functional analysis of hepatitis B virus reactivating in hepatitis B surface antigen-negative individuals. Hepatology 2005;42:93-103. [Crossref] [PubMed]
- Candotti D, Lin CK, Belkhiri D, et al. Occult hepatitis B infection in blood donors from South East Asia: molecular characterization and potential mechanisms of occurrence. Gut 2012;61:1744-53. [Crossref] [PubMed]
- Lewandowska MA. The missing puzzle piece: splicing mutations. Int J Clin Exp Pathol 2013;6:2675-82. [PubMed]
- Wang Z, Burge CB. Splicing regulation: from a parts list of regulatory elements to an integrated splicing code. RNA 2008;14:802-13. [Crossref] [PubMed]
- Guang S, Mertz JE. Pre-mRNA processing enhancer (PPE) elements from intronless genes play additional roles in mRNA biogenesis than do ones from intron-containing genes. Nucl Acids Res 2005;33:2215-26. [Crossref] [PubMed]
- Chi B, Wang K, Du Y, et al. A sub-element in PRE enhances nuclear export of intronless mRNAs by recruiting the TREX complex via ZC3H18. Nucl Acids Res 2014;42:7305-18. [Crossref] [PubMed]
- Smith GJ, Donello JE, Lück R, et al. The hepatitis B virus post-transcriptional regulatory element contains two conserved RNA stem-loops which are required for function. Nucleic Acids Res 1998;26:4818-27. [Crossref] [PubMed]
- Schwalbe M, Ohlenschläger O, Marchanka A, et al. Solution structure of stem-loop alpha of the hepatitis B virus post-transcriptional regulatory element. Nucleic Acids Res 2008;36:1681-9. [Crossref] [PubMed]
- Buratti E, Baralle FE. Influence of RNA secondary structure on the pre-mRNA splicing process. Mol Cell Biol 2004;24:10505-14. [Crossref] [PubMed]
- Loeb DD, Mack AA, Tian R. A secondary structure that contains the 5’ and 3’ splice sites suppresses splicing of duck hepatitis B virus pregenomic RNA. J Virol 2002;76:10195-202. [Crossref] [PubMed]
- Heise T, Sommer G, Reumann K, et al. The hepatitis B virus PRE contains a splicing regulatory element. Nucleic Acids Res 2006;34:353-63. [Crossref] [PubMed]
- Huang C, Xie MH, Liu W, et al. A structured RNA in hepatitis B virus post-transcriptional regulatory element represses alternative splicing in a sequence-independent and position-dependent manner. FEBS J 2011;278:1533-46. [Crossref] [PubMed]
- zu Putlitz J, Tong S, Wands JR. A short region in the genome of hepatitis B virus is critical for maintenance of high transcript levels. Virology 1999;254:245-56. [Crossref] [PubMed]
- Chowdhury JB, Roy D, Ghosh S. Identification of a unique splicing regulatory cluster in hepatitis B virus pregenomic RNA. FEBS Lett 2011;585:3348-53. [Crossref] [PubMed]
- Gozani O, Patton JG, Reed R. A novel set of spliceosome-associated proteins and the essential splicing factor PSF bind stably to pre-mRNA prior to catalytic step II of the splicing reaction. EMBO J 1994;13:3356-67. [PubMed]
- Zang WQ, Li B, Huang PY, et al. Role of polypyrimidine tract binding protein in the function of the hepatitis B virus posttranscriptional regulatory element. J Virol 2001;75:10779-86. [Crossref] [PubMed]
- Roy D, Chowdhury JB, Ghosh S. Polypyrimidine tract binding protein (PTB) associates with intronic and exonic domains to squelch nuclear export of unspliced RNA. FEBS Lett 2013;587:3802-7. [Crossref] [PubMed]
- Zang WQ, Fieno AM, Grant RA, et al. Identification of glyceraldehyde-3-phosphate dehydrogenase as a cellular protein that binds to the hepatitis b virus posttranscriptional regulatory element. Virology 1998;248:46-52. [Crossref] [PubMed]
- Beames B, Lanford RE. Carboxy_terminal truncations of the HBV core protein affect capsid formation and the apparent size of encapsidated HBV RNA. Virology 1993;194:597-607. [Crossref] [PubMed]
- Li HC, Huang EY, Su PY, et al. Nuclear export and import of human hepatitis B virus capsid protein and particles. PLoS Pathog 2010;6:e1001162 [Crossref] [PubMed]
- Köck J, Nassal M, Deres K, et al. Hepatitis B virus nucleocapsids formed by carboxy-terminally mutated core proteins contains spliced viral genomes but lack full-size DNA. J Virol 2004;78:13812-8. [Crossref] [PubMed]
- Le Pogam S, Chua PK, Newman M, et al. Exposure of RNA templates and encapsidation of spliced viral RNA are influenced by the arginine-rich domain of human hepatitis B virus core antigen (HBcAg 165-173). J Virol 2005;79:1871-87. [Crossref] [PubMed]
- Sheen IS, Tsou YK, Lin SM, et al. Nuclear HBcAg and histology activity index as independent predictors of the expression of singly spliced HBV-RNA. J Viral Hepat 2007;14:70-4. [Crossref] [PubMed]
- Mabit H, Knaust A, Breiner KM, et al. Nuclear localization of the duck hepatitis B virus capsid protein: detection and functional implications of distinct subnuclear bodies in a compartment associated with RNA synthesis and maturation. J Virol 2003;77:2157-64. [Crossref] [PubMed]
- Huang HL, Jeng KS, Hu CP, et al. Identification and characterization of a structural protein of hepatitis B virus: a polymerase and surface fusion protein encoded by a spliced RNA. Virology 2000;275:398-410. [Crossref] [PubMed]
- Park GS, Kim HY, Shin HS, et al. Modulation of hepatitis B virus replication by expression of polymerase-surface fusion protein through splicing: implications for viral persistence. Virus Res 2008;136:166-74. [Crossref] [PubMed]
- Obert S, Zachmann-Brand B, Deindl E, et al. A spliced hepadnavirus RNA that is essential for virus replication. EMBO J 1996;15:2565-74. [PubMed]
- Chen WN, Chen JY, Lin WS, et al. Hepatitis B doubly spliced protein, generated by a 2.2 kb doubly spliced hepatitis B virus DNA, is a pleiotropic activator protein mediating its effect via activator protein-1- and CCAAT/enhancer-binding protein-binding sites. J Gen Virol 2010;91:2592-600. [Crossref] [PubMed]
- Soussan P, Garreau F, Zylberberg H, et al. In vivo expression of a new hepatitis B virus protein encoded by a spliced RNA. J Clin Invest 2000;105:55-60. [Crossref] [PubMed]
- Bayard F, Godon O, Nalpas B, et al. T-cell responses to hepatitis B splice-generated protein of hepatitis B virus and inflammatory cytokines/chemokines in chronic hepatitis B patients. ANRS study: HB EP 02 HBSP-FIBRO. J Viral Hepat 2012;19:872-80. [Crossref] [PubMed]
- Lee GH, Wasser S, Lim SG. Hepatitis B pregenomic RNA splicing-The products, the regulatory mechanisms and its biological significance. Virus Res 2008;136:1-7. [Crossref] [PubMed]
- Lu YW, Tan TL, Chan V, et al. The HBSP gene is expressed during HBV replication, and its coded BH3-containing spliced viral protein induces apoptosis in HepG2 cells. Biochem Biophys Res Commun 2006;351:64-70. [Crossref] [PubMed]
- Soussan P, Tuveri R, Nalpas B, et al. The expression of hepatitis B spliced protein (HBSP) encoded by a spliced hepatitis B virus RNA is associated with viral replication and liver fibrosis. J Hepatol 2003;38:343-8. [Crossref] [PubMed]
- Chen JY, Chen WN, Liu LL, et al. Hepatitis B spliced protein (HBSP) generated by a spliced hepatitis B virus RNA participates in abnormality of fibrin formation and functions by binding to fibrinogen gamma chain. J Med Virol 2010;82:2019-26. [Crossref] [PubMed]
- Chen WN, Chen JY, Jiao BY, et al. Interaction of the hepatitis B spliced protein with cathepsin B promotes hepatoma cell migration and invasion. J Virol 2012;86:13533-41. [Crossref] [PubMed]
- Pol JG, Lekbaby B, Redelsperger F, et al. Alternative splicing-regulated protein of hepatitis B virus hacks the TNF-α-stimulated signaling pathways and limits the extent of liver inflammation. FASEB J 2015;29:1879-89. [Crossref] [PubMed]
- Chen J, Wu M, Wang F, et al. Hepatitis B virus spliced variants are associated with an impaired response to interferon therapy. Sci Rep 2015;5:16459. [Crossref] [PubMed]
- Wang YL, Liou GG, Lin CH, et al. The inhibitory effect of the hepatitis B virus singly-spliced RNA-encoded p21.5 protein on HBV nucleocapside formation. PLoS One 2015;10:e0119625 [Crossref] [PubMed]
- Louro R, Smirnova AS, Verjovski-Almeida S. Long intronic noncoding RNA transcription: expression noise or expression choice? Genomics 2009;93:291-8. [Crossref] [PubMed]
- Li C, Hu J, Hao J, et al. Competitive virus and host RNAs: the interplay of a hidden virus and host interaction. Protein Cell 2014;5:348-56. [Crossref] [PubMed]
- Tsai KN, Chong CL, Chou YC, et al. Doubly spliced RNA of hepatitis B virus suppresses viral transcription via TATA-binding protein and induces stress granule assembly. J Virol 2015;89:11406-19. [Crossref] [PubMed]
- Cox LE, Arslan O, Allain JP. Characterization of hepatitis B virus in Turkish blood donors, and the prevalence of the SP1 splice variant. J Med Virol 2011;83:1321-5. [Crossref] [PubMed]
- Ma ZM, Lin X, Wang YX, et al. A double-spliced defective hepatitis B virus genome derived from hepatocellular carcinoma tissue enhanced replication of full-length virus. J Med Virol 2009;81:230-7. [Crossref] [PubMed]
- Ogston CW, Razman DG. Spliced RNA of woodchuck hepatitis virus. Virology 1992;189:245-52. [Crossref] [PubMed]
- Yuan TT, Lin MH, Chen DS, et al. A defective interference-like phenomenon of human hepatitis B virus in chronic carriers. J Virol 1998;72:578-84. [PubMed]
- Märschenz S, Endres AS, Brinckmann A, et al. Functional analysis of complex hepatitis B virus variants associated with development of liver cirrhosis. Gastroenterology 2006;131:765-80. [Crossref] [PubMed]
- Lin X, Wen Y, Wan D, et al. Structural and functional analysis of 2.2 kb spliced variant of hepatitis B virus genomes isolated from liver tissues from hepatocellular carcinoma patients. Zhonghua Shi Yan He Lin Chuang Bing Du Xue Za Zhi 2002;16:11-5. [PubMed]
- Soussan P, Pol J, Garreau F, et al. Expression of defective hepatitis B virus particles derived from singly spliced RNA is related to liver disease. J Infect Dis 2008;198:218-25. [Crossref] [PubMed]
- Kew MC. Hepatitis B virus x protein in the pathogenesis of hepatitis B virus-induced hepatocellular carcinoma. J Gastroenterol Hepatol 2011;26:144-52. [Crossref] [PubMed]
- Chiu YT, Wong JK, Choi SW, et al. Novel pre-mRNA splicing of intronically integrated HBV generates oncogenic chimera in hepatocellular carcinoma. J Hepatol 2016;64:1256-64. [Crossref] [PubMed]
- Allain JP, Candotti D. Diagnostic algorithm for HBV safe transfusion. Blood Transfusion 2009;7:174-82. [PubMed]
- Warf MB, Berglund JA. The role of RNA structure in regulating pre-mRNA splicing. Trends Biochem Sci 2010;35:169-78. [Crossref] [PubMed]
- Huang FY, Wong DK, Seto WK, et al. Sequence variations of full-length hepatitis B virus genomes in Chinese patients with HBsAg-negative hepatitis B infection. PLoS One 2014;9:e99028 [Crossref] [PubMed]
- Chemin I, Alain S, Margeridon S, et al. What is really ongoing during occult HBV reactivation? Hepatology 2006;43:195-author reply 195-6. [Crossref] [PubMed]

蒋祺
河南大学医学院免疫学硕士研究生学历,输血技术中级职称,开封市检验协会委员,2015年至今任核酸实验室主任。长期从事血液分子生物学研究,研究生期间参与2项国家自然科学基金研究工作。2016年,在中国医师协会和输血医师分会联合举办的“迎接新常态,勇做输血领军人”全国人才选拔活动中,入选“全国输血医学人才库”。在国家级刊物中发表多篇学术论文。(更新时间:2021/8/26)
(本译文仅供学术交流,实际内容请以英文原文为准。)
Cite this article as: Candotti D, Allain JP. Biological and clinical significance of hepatitis B virus RNA splicing: an update. Ann Blood 2017;2:6.